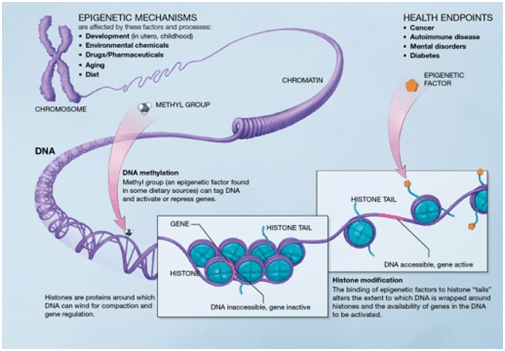
La epigenética es un campo de investigación que ha experimentado un ‘boom’ en los últimos años gracias al avance en las técnicas que facilitan su estudio. Se centra en los cambios producidos en el DNA e histonas que modifican la estructura de la cromatina sin afectar a la secuencia de nucleótidos pero produciendo cambios en la expresión génica. Existen diversas modificaciones de las histonas: metilación, acetilación, ADP-ribosilación, ubiquitinación, citrulación y fosforilación.
La metilación de DNA es una de las modificaciones más importantes de la cromatina ya que desempeña un papel esencial en el silenciamiento o la activación de la expresión génica, afectando a todos los procesos celulares. Cambios en los patrones de metilación aparecen en las primeras etapas del desarrollo del cáncer y otras enfermedades.
Existen diversas técnicas desarrolladas para la detección de la metilación del DNA: digestión por bisulfito, restricción enzimática, e inmuprecipitación de cromatina (ChIP). Nos vamos a centrar en la inmunoprecipitación de cromatina.
Inmunoprecipitación de cromatina (ChIP)
La inmunoprecipitación de cromatina (ChIP) es una técnica utilizada para el estudio de la unión de proteínas a regiones específicas de DNA genómico. Gracias a esta técnica podemos tener una ‘imagen’ de las interacciones proteína-DNA en el núcleo celular. Estas interacciones juegan un papel fundamental en la regulación génica.
Las mayores debilidades de esta técnica han sido: la necesidad de muestras grandes debido a las pérdidas de muestra en los lavados, una baja señal general unida al ruido y una reproducibilidad bastante complicada.
Cada vez más estudios muestran cómo la unión de estas proteínas, ya sean factores de transcripción o histonas modificadas, se unen a una región determinada del DNA en células o tejidos produciendo diferentes patrones de metilación en función de la enfermedad que sea.
En un experimento ChIP, los fragmentos de cromatina (el complejo DNA-proteína) son tratados de tal forma que se retienen las interacciones específicas DNA -proteína. Tras esto, la cromatina se extrae y se fragmenta en pequeños fragmentos de 100-500 pares de bases a través de sonicación o digestión enzimática. Estos fragmentos son inmunoprecipitados de manera selectiva utilizando anticuerpos contra la proteína de interés. A continuación se separa el DNA y los componentes proteicos.
Chromatrap extrae la cromatina más fácil y más eficientemente
¿Cómo? Los kits de Chromatrap utilizan tanto columnas como microplacas con filtro en la base que contienen discos de un polímero poroso unido covalentemente a Proteínas A o G. Este formato patentado es único. Durante el ensayo, el complejo cromatina/anticuerpo es retenido selectivamente por el disco. Para obtener el DNA enriquecido de interés, tan sólo son necesarios 3 lavados y una elución. 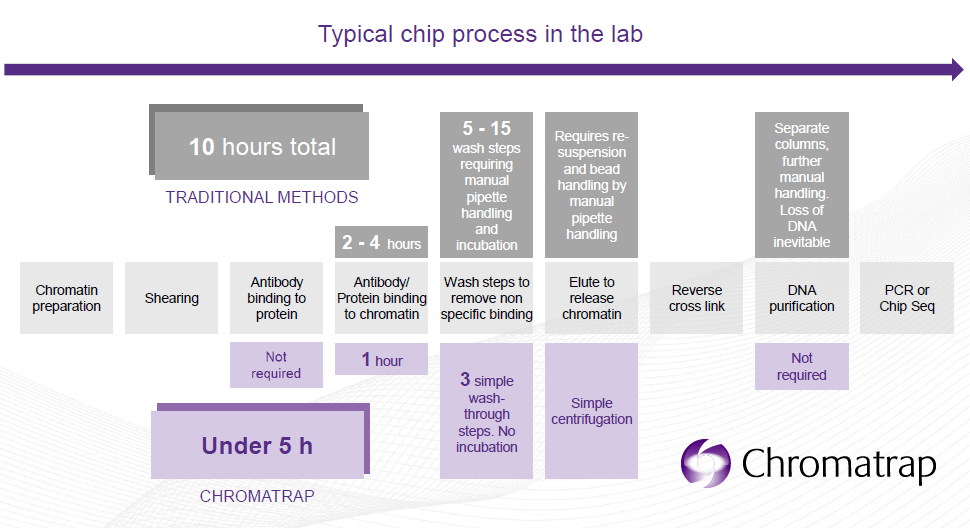
Este proceso desarrollado por Chromatrap es el más eficiente a nivel de laboratorio y presenta las siguientes ventajas:
- ChIP en menos de 5 horas: desde la preparación de la cromatina hasta la qPCR.
- Menos manipulación de la muestra, elimina la pérdida de muestra tras múltiples pipeteos.
- Formatos para ensayos individuales y de placas de 96 pocillos.
- Optimizado para muestras de 1000ng ya que las muestras más pequeñas dan mejores resultados en qPCR.
- Permite hacer más ensayos IP por muestra.
En el siguiente vídeo se explica el proceso:
¿Tienes dudas?
Si no te queda claro del todo cómo funciona esta tecnología, o quieres que te ayudemos a configurar tu ensayo, nuestro departamento técnico de especialistas con amplia trayectoria en investigación (todos PhD) te pueden echar una mano, por mail (tecnic@labclinics.com), por tlf +34.934464700 o presencial. Contáctanos y estaremos encantados de poder ayudarte!
Si quieres mantenerte informad@ sobre esta y otras noticias, apúntate a nuestra newsletter aquí
Referencias:
García R, Ayala PA, Perdomo SP. Epigenética: definición, bases moleculares e implicaciones en la salud y en la evolución humana. Rev. Cienc. Salud 2012; 10 (1):59-71.






Deja tu respuesta