En la entrada de este mes, te describimos los cinco errores de principiante a la hora de hacer una PCR cuantitativa (qPCR; quantitative PCR) y la mejor manera de evitarlos. Ya sea porque es la primera vez que realizas una qPCR o bien porque simplemente quieres asegurarte de maximizar tus posibilidades de éxito, aquí te mostramos algunas normas básicas que debes tener en cuenta.
- 1. Poca calidad del RNA
- 2. Escoger el tipo de detección
- 3. No utilizar una mastermix y no organizar tus experimentos
- 4. Escatimar en el número de controles
- 5. No validar tus resultados con un gen de referencia
1. Poca calidad del RNA
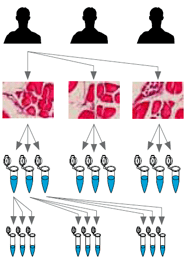 Este es posiblemente el paso más crítico para preparar de forma exitosa nuestro cDNA y poder realizar una qPCR eficiente y reproducible. El RNA es un material extremadamente sensible a la degradación por RNasas y debe manejarse con cuidado durante los pasos de aislamiento de ácidos nucleicos.
Este es posiblemente el paso más crítico para preparar de forma exitosa nuestro cDNA y poder realizar una qPCR eficiente y reproducible. El RNA es un material extremadamente sensible a la degradación por RNasas y debe manejarse con cuidado durante los pasos de aislamiento de ácidos nucleicos.
El RNA degradado o contaminado puede afectar negativamente la eficiencia y rendimiento de nuestra RT-qPCR. Pipetas, puntas o incluso la propia poyata del laboratorio, pueden ser foco de RNAsas y deben estar extremadamente limpio para utilizarlos con el propósito de aislar RNA.
Una vez extraído nuestro RNA, debemos evaluar su pureza. Una opción es determinarlo mediante espectrofotometría. Además de la concentración, nos debemos fijar en la relación de absorbancia entre 260 y 280 nm (A260/A280). Esta relación debe estar en el rango de 1.8-2.0. Si es inferior, puede sugerir la contaminación de la muestra por fenol o proteínas.
2. Escoger el tipo de detección
Cada PCR en tiempo real contiene una molécula indicadora de fluorescencia, bien sea una sonda (tipo Taqman®, Molecular Beacons o Scorpions™ Probes). A medida que aumenta la cantidad de amplicón deseado, también lo hace la cantidad de fluorescencia emitida por el fluoróforo.
Existen dos tipos de ensayo para realizar estudios de expresión génica mediante PCR en tiempo real:
- Ensayos basados en sondas Taqman® (también llamados ensayos del tipo nucleasa 5′ fluorogénico)
- Ensayos basados en SYBR® Green dye.
En el siguiente cuadro te indicamos en qué situación es mejor utilizar uno o el otro.
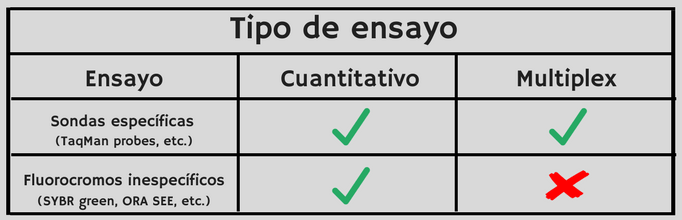 Cada tipo de ensayo tiene sus ventajas y limitaciones. Por ejemplo, la tecnología basada en sondas permite realizar PCR multiplex. Sin embargo, si tu prioridad es obtener una alta sensibilidad, la tecnología basada en green dyes puede ser la más adecuada. En este cuadro, te mostramos las ventajas y desventajas de cada uno de los sistemas.
Cada tipo de ensayo tiene sus ventajas y limitaciones. Por ejemplo, la tecnología basada en sondas permite realizar PCR multiplex. Sin embargo, si tu prioridad es obtener una alta sensibilidad, la tecnología basada en green dyes puede ser la más adecuada. En este cuadro, te mostramos las ventajas y desventajas de cada uno de los sistemas.

3. No utilizar una mastermix y no organizar tus experimentos

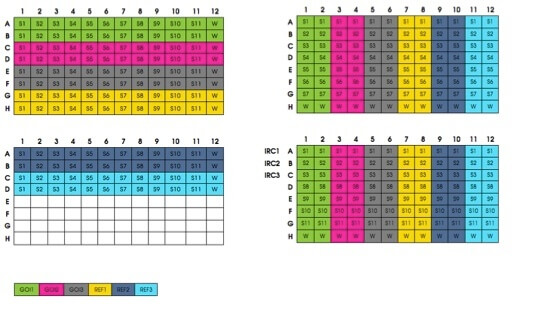
El uso de una mastermix minimiza la variabilidad experimental y, por lo tanto, mejora la reproducibilidad al reducir las variaciones pocillo a pocillo y de muestra a muestra. Del mismo modo, es fundamental mantenerse organizado en el diseño y la ejecución de nuestros experimentos de qPCR. Por ejemplo, es útil realizar una tabla que incluya los primers y las muestras para saber qué va y a dónde va. No hay nada peor que distraerse y olvidarse de qué pocillo tiene y no ha sido pipeteado. Algunos investigadores eligen su diseño en un patrón, por ejemplo los primers ordenados alfabéticamente.
De esta manera, al cargar la muestra se pueden hacer coincidir las puntas con los pocillos a la manera del juego «Hundir la Flota» (es decir, punta C7 a C7, etc…). No importa la estrategia de pipeteo, pero no te olvides de hacer un spin a tu placa antes de meterla en el termociclador para evitar que cualquier líquido quede en las paredes de los pocillos.
4. Escatimar en el número de controles
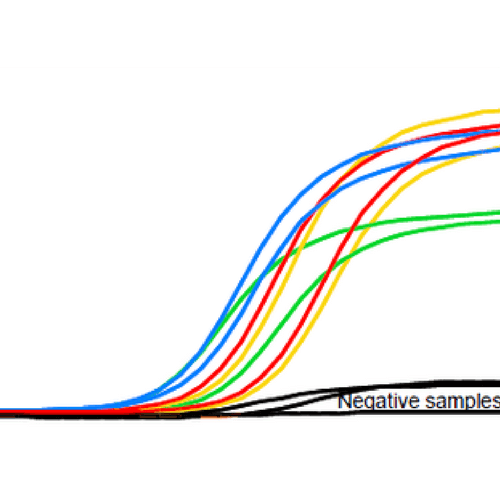
Es importante incluir los controles apropiados en la configuración de nuestros experimentos de RT-qPCR. Estos, deben incluir un control negativo que no contenga RNA o cDNA y reemplazar el volumen con agua (libre de nucleasas), es el denominado «control sin muestra«. Este control, permite detectar contaminación cruzada de superficies o reactivos (es decir, problemas de contaminación en mastermix o primers) así como la formación de primer-dimers. Por lo tanto, si observas amplificación en este pocillo, deberías correr un gel de agarosa para averiguar de qué se trata. Otro control negativo es el siguiente: una muestra negativa a la cual no se le ha añadido la retro-transcriptasa inversa, de tal forma que no se ha formado el cDNA. Si se detecta producto, este resultado sugiere contaminación de la muestra por DNA exógeno.
5. No validar los resultados mediante un gen de referencia

La amplificación de un gen control (o de referencia) permite la normalización de la expresión del gen diana mediante la comparación de los valores de Ct. Muchas veces, esto puede explicar las diferencias en la cantidad o calidad del material de partida (RNA o cDNA), así como las diferencias en los métodos de preparación de RNA o la síntesis de cDNA. Un gen de referencia veraz es aquel cuyo nivel de expresión no se ve afectado por sus variables o intervenciones experimentales y además no difiere entre los estados fisiológicos relevantes de sus condiciones de muestra.
¿Tienes dudas?
Si no te queda claro del todo cómo funciona esta tecnología, o quieres que te ayudemos a configurar tu ensayo, nuestro departamento técnico de especialistas, con amplia trayectoria en investigación (todos PhD), te pueden echar una mano: por mail (tecnic@labclinics.com), por tlf +34.934464700 o de forma presencial. Contáctanos y estaremos encantados de poder ayudarte!







Deja tu respuesta